研究室に配属されたばかりの新入生や、これからRで統計分析を始めたいと思っている方へ向けて、【R講座】では、RとRStudioの基本的な使い方から統計手法の選び方、基本的なデータ分析方法を解説しています。特にRが初めての方でも安心して学べるように、RStudioのクリック操作も紹介していきます。実際のコード例を交えながら進めるので、これからの研究やデータ分析に、役立てていただけたら嬉しいです。
今回の講座では、Rを使用してt検定を行う方法について解説しています。
t検定とは
t検定は、2つのグループの平均値に統計的に有意な差があるかどうかを調べる手法です。
前提条件
t検定を実施する際には以下の条件を確認します。
- 対応の有無:
- 対応のあるデータ:同じ対象の前後比較(例:同じ人のダイエット前後の体重)
- 対応のないデータ:異なる対象の比較(例:異なる人々のグループ間比較)
- 正規性: データが正規分布に従っているか。
- 等分散性:2つの群のデータが等分散かどうか。
- 群数:比較する群が2つであること。
- データ尺度:データが間隔尺度または比率尺度であること。
仮説の設定
t検定を実施する前に、以下の仮説を設定します。
- 帰無仮説(H0):2つの群の平均に差はない。
- 対立仮説(H1):2つの群の平均に差がある。
t検定の種類
- ステューデントのt検定:2群の分散が等しいと仮定
- ウェルチのt検定:2群の分散が等しいと仮定しない場合に使用
- 対応のあるt検定:同一対象から得られた2つの関連する標本の比較
t検定の関数と引数オプション
Rでt検定を行うには、t.test()関数を使用します。引数を変更することで、ステューデントのt検定・ウェルチのt検定・対応のあるt検定を実行することができます。
この関数は次の引数が設定されています。
# 2つのベクトルを使う方法
t.test(x, y = NULL,
alternative = c("two.sided", "less", "greater"),
mu = 0, paired = FALSE, var.equal = FALSE,
conf.level = 0.95, ...)
# データフレームを使う方法
t.test(formula, data, subset, na.action, ...)- x: 空でない数値ベクトルを指定します。
- y: 任意の空でない数値ベクトルを指定します。指定しない場合、1標本のt検定が行われます。
- alternative: 対立仮説を指定する文字列。"two.sided"(両側検定、デフォルト)、"greater"(右片側検定)、または "less"(左片側検定)を指定します。頭文字だけでも指定可能です。
- mu: 帰無仮説での平均値を指定する数値。2標本検定の場合は、平均値の差を指定します。
- paired: 対応のあるt検定を行うかどうかを指定する論理値。TRUEの場合、対応のあるt検定が行われます。
- var.equal: 2つの分散が等しいと仮定するかどうかを指定する論理値。TRUEの場合、2つの群のデータを結合して1つの共通分散を計算し、それを用いて検定を行います。FALSEの場合、ウェルチまたはサタースウェイトの自由度近似を使用します。
- conf.level: 信頼区間の信頼水準を指定する数値。デフォルトは95%です。
- formula: lhs ~ rhs形式の式。lhsは数値変数を指定し、rhsは1標本の場合は1を指定します。対応のある検定の場合や2水準の因子に基づく検定の場合、rhsに対応するグループを指定します。
- data: formulaで指定された変数を含むオプションのデータフレームまたは行列。
- subset: 使用する観測値のサブセットを指定する任意のベクトル。
- na.action: データにNAが含まれる場合の処理方法を指定する関数。デフォルトはgetOption("na.action")です。
簡単にまとめると、t.test()関数では、次の2通りの方法を使うことができます。
- ベクトル型の場合:
t.test(ベクトル1,ベクトル2) - フォーミュラ型の場合:
t.test(目的変数 ~ 説明変数)
注意点が色々あるので下にメモしておきます。
ステューデントとウェルチのt検定は、ベクトル型・フォーミュラ型のどちらでも検定を実行できますが、対応のあるt検定はフォーミュラ型で実行します。
フォーミュラ型で引数オプションのpaired = TRUEを指定すると次のエラーが表示されます。
t.test(data$`Height (cm)` ~ data$`Temperature (°C)`, paired = TRUE)Error in t.test.formula(data$`Height (cm)` ~ data$`Temperature (°C)`, :
cannot use 'paired' in formula method分析の実践
次のステップでデータを分析していきます。
- STEP 1データの読み込み
csvファイルからデータを読み込みます。
- STEP 2データの型変換(キャスト)
読み込んだデータの要因データを、Factor型に変更します。
- STEP 3t検定の実行
t.test()関数でt検定を実行します。 - STEP 4結果の出力
計算された結果がコンソールペインに出力されます。
使用するデータ
今回は、次のcsvファイルを使用して説明します。
t.test()関数のフォーミュラ型に対応したデータ入力法方にしています。
このcsvファイルには次のデータが含まれています。
| Temperature (°C) | Height (cm) | Weight (g) |
|---|---|---|
| 25 | 9.881208 | 57.412763 |
| 25 | 10.197684 | 50.685115 |
| 25 | 8.931307 | 46.762492 |
| 25 | 9.196787 | 39.134970 |
| 25 | 8.886235 | 39.840711 |
| 25 | 11.580092 | 42.322098 |
| 25 | 11.497819 | 38.802799 |
| 25 | 10.262645 | 45.518258 |
| 25 | 8.767099 | 54.717364 |
| 25 | 9.996276 | 38.195093 |
| 30 | 16.511672 | 114.702570 |
| 30 | 14.524302 | 86.885794 |
| 30 | 15.797916 | 99.034751 |
| 30 | 14.025997 | 123.697199 |
| 30 | 15.689373 | 108.906265 |
| 30 | 14.044161 | 97.478168 |
| 30 | 13.768293 | 91.342362 |
| 30 | 14.043108 | 105.825860 |
| 30 | 14.130217 | 99.874707 |
| 30 | 14.089319 | 96.251452 |
データの読み込み
まずは、次のコードを使って、オブジェクト「data」にread.csv()関数でcsvファイルのデータを代入します。
# データの読み込み
data <- read.csv(file.choose(),
check.names = F)csvファイルの読み込みでは、パスを指定することもできますが、今回はfile.choose関数でファイルを選択する方法をとっています。
パスを指定する方法についてはこちらで紹介しています。
確認のために、コンソールペインにdataと入力すると、次の表示が出力されます。
> data
Temperature (°C) Height (cm) Weight (g)
1 25 9.881208 57.41276
2 25 10.197684 50.68512
3 25 8.931307 46.76249
4 25 9.196787 39.13497
5 25 8.886235 39.84071
6 25 11.580092 42.32210
7 25 11.497819 38.80280
8 25 10.262645 45.51826
9 25 8.767099 54.71736
10 25 9.996276 38.19509
11 30 16.511672 114.70257
12 30 14.524302 86.88579
13 30 15.797916 99.03475
14 30 14.025997 123.69720
15 30 15.689373 108.90626
16 30 14.044161 97.47817
17 30 13.768293 91.34236
18 30 14.043108 105.82586
19 30 14.130217 99.87471
20 30 14.089319 96.25145データの整理
次に、データの因子変換をします。
Rでは説明変数をFactor型のデータとします。今回の分析ではTemperature (°C)を説明変数とするので、as.factor()関数でデータ型をinteger型からFactor型に変更します。
# 説明変数をデータ型をFactor型に変更
data$`Temperature (°C)` <- as.factor(data$`Temperature (°C)`)実行結果は、Environmentタブで確認できます。Temperature (°C)のデータ型がinteger型(左図)からFactor型(右図)に変わります。
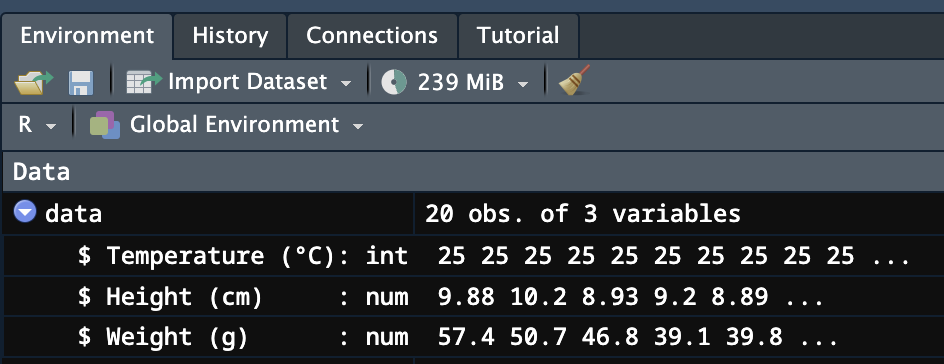
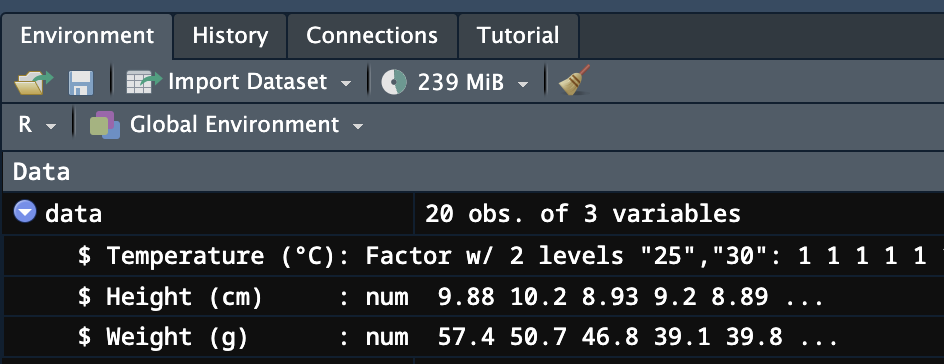
ここで、バッククォート「`」について、少しだけ補足します。
R言語においてバッククォート「 ` 」で文字列を挟むと、通常は予約語や空白、特殊文字を含む名前を使うことができます。これにより、通常は無効な識別子として扱われる文字列を有効なオブジェクト名として扱うことができます。
ヘッダー名であるHeight (cm)は、スペース「 」や括弧「()」が含まれているため、そのままオブジェクト名として使用できませんが、バッククォートを使うことでオブジェクト名として使用できます。
t検定の実行
次に、以下のコードでウェルチのt検定を実行します。
(引数にvar.equal = TRUEを追加することで、ステューデントのt検定を実行できます。)
# ウェルチのt検定を実行
t.test(data$`Height (cm)` ~ data$`Temperature (°C)`)入力法方はフォーミュラ型で、目的変数はHeight (cm)のデータ、説明変数はTemperature (°C)としています。目的変数を変更すれば、Weight (g)のデータを検定できます。
結果の出力
検定を実行した結果は、コンソールペインに表示されます。
Welch Two Sample t-test
data: data$`Height (cm)` by data$`Temperature (°C)`
t = -10.691, df = 17.945, p-value = 3.266e-09
alternative hypothesis: true difference in means between group 25 and group 30 is not equal to 0
95 percent confidence interval:
-5.674898 -3.810543
sample estimates:
mean in group 25 mean in group 30
9.919715 14.662436 有意水準が5%とするとき、p値(p-value)が0.05より小さい場合、帰無仮説を棄却し、2つの群の平均に有意な差があると結論づけます。
また、95%信頼区間(95 percent confidence interval)は、真の平均値の差が95%の確率で含まれている範囲を示しています。(この区間に0が含まれる場合は、平均値の差が0の可能性があります)
今回の結果から、p値が3.266e-09であり、95%信頼区間が-5.674898 から -3.810543である(0が含まれない)ため、2つの群の平均に有意な差があると結論できます。
まとめ
今回は、Rを使用してt検定を行う方法について解説しました。
最後に、コードをまとめておきます。
# データの読み込み
data <- read.csv(file.choose(),
check.names = F)
# 説明変数をデータ型をFactor型に変更
data$`Temperature (°C)` <- as.factor(data$`Temperature (°C)`)
# ウェルチのt検定を実行
t.test(data$`Height (cm)` ~ data$`Temperature (°C)`)次回は、分散分析の方法について紹介します。




